Analyse a T1-weighted histogram to estimate grey/white statistics
Cette procédure analyse la forme de l'histogramme d'une IRM pondérée en T1 pour en déduire une estimation des moyennes et des écart types des matières grises et blanches. La multiplicité des constructeurs de scanners et des séquences utilisées, ainsi que la variabilité anatomique et les biais spatiaux qui entachent les images, rendent cette estimation difficile dans le cas général. En effet, la forme des histogrammes est très variées, ils contiennent généralement entre trois et cinq pics significatifs, voire plus en cas de problèmes d'échantillonage.
Les seules propriétés sur lesquels peut s'appuyer cette procédure sont donc:Malheureusement, nous avons souvent été également confrontés à des images cliniques dotées d'un très mauvais contraste. Dans de telles situations, les tissus correspondant aux matières grises et blanches ne correspondent qu'à un pic dans l'histogramme. Heureusement, les dérivées de l'histogramme permettent néanmoins de déceler l'existence de deux classes de tissus.
- l'existence systématique d'un pic pour le fond, le gris et le blanc, apparaissant dans cet ordre;
- le fait qu'il s'agit des pics correspondant aux plus grandes plages de voxels dans les cas non pathologiques. Cette propriété permet a priori de les distinguer des pics parfois induits par le liquide céphalo rachidien et la graisse.
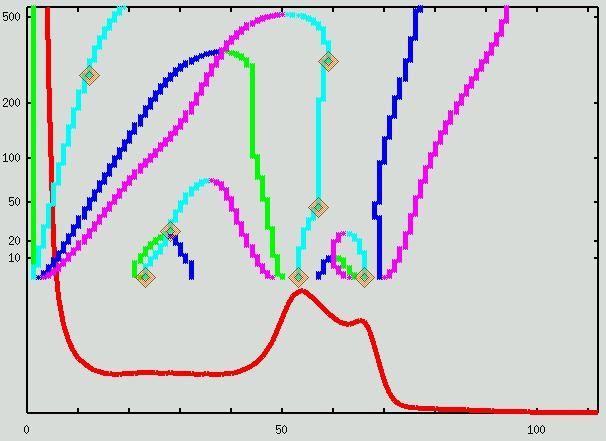
L'heuristique qui réalise l'analyse d'un histogramme quelconque s'appuie sur la notion d'échelle. L'idée consiste à lisser l'histogramme jusqu'à ce que seuls les pics les plus importants restent visibles. Ce lissage correspond en fait à appliquer l'équation de la chaleur à l'histogramme (ce qui équivaut à des lissage gaussiens). L'évolution de l'histogramme dans l'espace des échelles est alors interprétée à partir du suivi des extremas de certaines dérivées. Ces extremas parcourent des trajectoires parfois relativement esthétiques qui nous permettent de déméler les choses dans la plupart des situations...
Lorsqu'un pic est isolé, il est doté d'une signature caractéristique dans le monde des trajectoires des extremas:
Lorsque deux pics se rapprochent au cours du lissage, des trajectoires disparaissent peu à peu à travers des phénomène d'extinction dont la syntaxe est systématique.
Les trajectoires propres à un pic donné sont démélées en s'appuyant sur ces règles de syntaxe. Il reste alors à extraire les signatures des pics qui nous intéressent:
L'ensemble des trajectoires:
Le résultat de l'analyse:
Des exemples variés d'analyses sont proposés ci dessous. Les pics gris et blanc, ainsi que le pic correspondant au liquide céphalo-rachidien, lorsqu'il existe, sont indiqués par une trajectoire cyan...
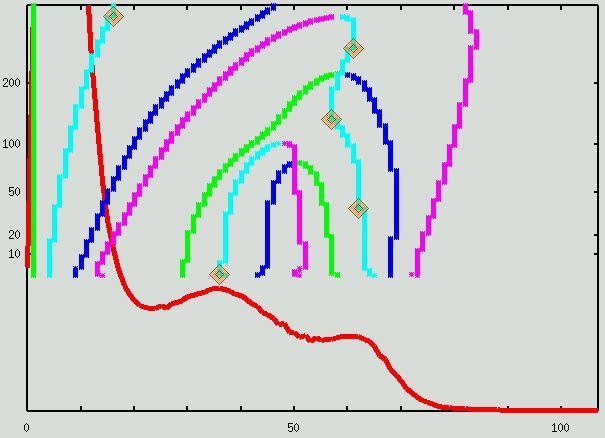
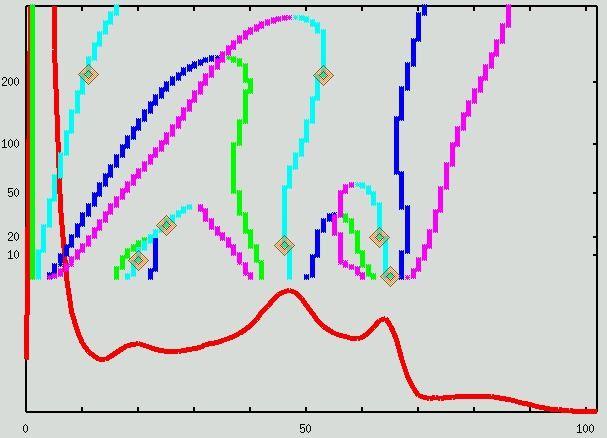
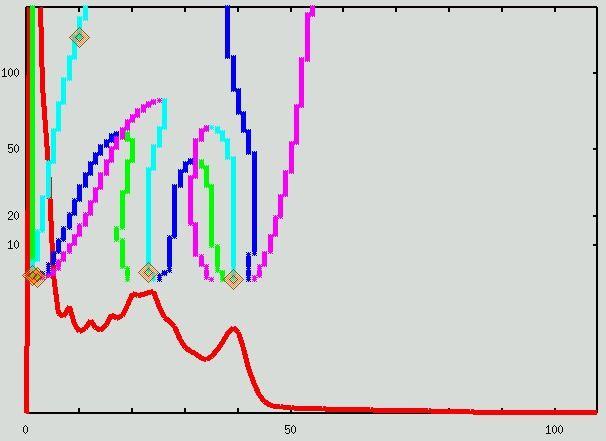
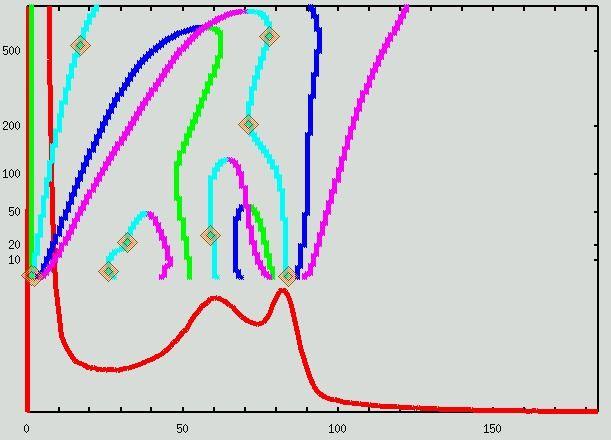
Pour plus d'information :
Robust brain segmentation using histogram scale-space analysis and mathematical morphology, J.-F. Mangin, O. Coulon, and V. Frouin MICCAI, MIT, LNCS-1496, Springer Verlag 1230-1241, 1998
Vous pouvez également essayer la commande ligne :
VipHistoAnalysis -help
mri_corrected: T1 MRI Bias Corrected ( input )
histo_analysis: Histo Analysis ( output )
text file
visu: Boolean ( input )gnuplot viewer
undersampling: Choice ( input )
Toolbox : Morphologist
User level : 2
Identifier :
VipHistoAnalysisSupported file formats :
mri_corrected :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Directory, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagehisto_analysis :Histo Analysis, Histo Analysis