Guide pour la correction d'une erreur de Ana Brain Mask From T1 MRI
Pour une raison ou pour une autre, la procédure Brain Mask From T1 MRI a échoué sur une de vos images. Avec un peu de chance, votre problème est répertorié dans ce qui suit, auquel cas vous devriez disposer d'une solution simple qui consistera à varier certains des paramètres pilotant le processus d'érosion. Des variantes utiles sont accessibles directement dans le menu variant de cette procédure. Une visualisation du résultat dans Anatomist est obtenue si vous sélectionnez Mask visualization dans le menu help.
Si rien ne marche, vous pourrez réaliser certaines corrections en dessinant dans Anatomist: Label volume editor (vous pouvez d'ailleurs utiliser un racourci vers ce traitement sous forme d'icone).
Si vous n'avez pas beaucoup de recul sur le processus mis en oeuvre pour extraire le masque du cerveau, essayez les variantes une à une jusqu'à obtenir satisfaction. Vous pouvez sélectionner la bonne variante en vous appuyant sur les exemples de problèmes qui suivent, classés par fréquence d'occurence. Si aucune variante ne vous satisfait vraiment, vous pourrez soit choisir la moins mauvaise, soit essayer d'accéder directement aux paramètres de la procédure. Les plus importants sont ajustables dans brainVISA à travers la brique Vip Get Brain, les autres sont accessibles dans la commande en ligne (VipGetBrain -help). Il devrait également être possible de corriger les problèmes directement à la main avec le module de dessin d'Anatomist, mais la documentation reste à développer...
Structures extra-cérébrales
Le masque contient des structures situées en dehors du cerveau:
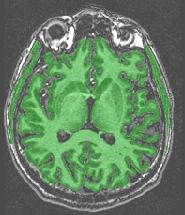
Verdict : le processus d'érosion sensé casser les connections entre le cerveau et l'extérieur (nerfs optiques, etc...) n'a pas été suffisamment important. Il s'agit d'un échec de la procédure visant à l'ajuster automatiquement. En général, les structures extracérébrales se situent sous le cervelet et le lobe temporal. Si seul le cervelet est en cause, vous pouvez en rester là dans la mesure ou la suite du pipeline vise à éliminer le cervelet. Si les hémisphères ont des excroissances, vous pourrez les éliminer à partir d'une des variantes permettant d'imposer la taille de l'érosion, en cherchant à minimiser cette taille. En cas de succès, vérifiez qu'une érosion trop importante n'a pas fait disparaître certains gyri.
Si les choix précédents ne vous ont pas satisfaits, il est possible que votre problème provienne d'un artefact de l'acquisition qui fait quasiment disparaître le liserai noir correspondant au crâne à certains endroits :
Auquel cas, la binarisation initiale qui essaie normalement de boucher les petites cavités résultant du bruit d'acquisition dans le cerveau doit être effectuée plus prudemment. En temps normal, ce traitement cherche à protéger les circonvolutions qui risquent d'être effacées par des érosions parasites venant de ces cavités. Si malheureusement, le liserai correspondant au crâne a été en partie effacé, le processus d'erosion ne peux plus séparer le cerveau des tissus externes. Essayez les choix suivant :"Standard/Robust ... without regularisation"
Une autre source d'erreur est liée au repliement fréquemment observé dans les premières et les dernières coupes de l'image (répétition de l'image en miroir, différences d'intensité, etc...). Ce type de problème peut engendrer un disfonctionnement complet de la procédure. Une solution "miracle" consiste à effacer les premières et les dernières coupes. Vous pouvez réessayer les différents choix ci-dessus en effaçant plus ou moins de coupes à l'aide des paramètres first_slice et last_slice. Vous pouvez même réessayer la procédure standard :
"Standard (iterative erosion from 2mm)"
Structures cérébrales érodées
Vous avez perdu quelques gyri en route...
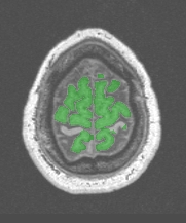
ce sont des choses qui arrivent. Si l'effet est trop important, essayez la variante de la procédure standard qui démarre avec une érosion de 1.5mm seulement:"Standard/Robust (iterative or fixed erosion from 1.5mm)"
Si ca ne suffit pas, voici quelques conseils à appliquer avec le traitement "Vip Get Brain" ou la commande ligne VipGetBrain.Ne soyez pas trop maniaques, ces morceaux de gyri peuvent être négligés dans la plupart des applications.
- Essayez de diminuer la taille de l'érosion (en particulier en cas d'atrophie);
- Vérifiez que les résultats de l'analyse d'histogramme sont corrects. En désespoir de cause, diminuez manuellement la moyenne du gris, ou augmentez son écart type;
- Augmentez le nombre d'itérations de la régularisation.
Résultats bizarres
La multitude d'ajouts permettant d'obtenir de bon résultats avec une grande variété d'images conduit parfois à des surprises. Par exemple, avec les procédures "Robust...", il arrive qu'une partie de la matière blanche du corps calleux et du tronc cérébral disparaisse:
La meilleure solution est alors de se rabattre sur une variante plus simple de la procédure:
"Fast (2mm erosion)"
"Fast (2.5mm erosion)"
"Fast (3mm erosion)"
Echec de l'analyse d'histogramme
Pour réaliser la binarisation initiale, c'est-à-dire une image noir et blanc qui indique les plages de tissu susceptibles d'appartenir au cerveau, une analyse automatique de l'histogramme est effectuée. Cette analyse recherche les deux bosses qui correspondent à la matière grise et à la matière blanche (Vip Histogram Analysis). Cette analyse est réussie si deux lignes bleu clair émergent du sommet de ces deux bosses comme dans l'exemple qui suit :
Vous pouvez accéder à cette visualisation en choisissant:
histogram analysis visualization
dans le menu Help (vous pouvez désactiver le processus de segmentation en choisissant Nothing dans le menu variant). Malheureusement, pour l'instant, cette visualisation bloque la brique de traitement, utilisez la avec parcimonie...
Si votre analyse d'histogramme n'est pas similaire à l'exemple (d'autres exemples ici), il est probable que la procédure de correction de biais qui précède cette analyse n'a pas atteint son objectif (cf ci-dessous), ou que votre image brute n'a pas un contraste gris/blanc suffisant.
En cas de désespoir profond, vous pouvez effectuer cette analyse manuellement en modifiant le fichier qui résulte de l'analyse automatique et qui sera utilisé par les procédure suivantes. Ce fichier, qui est doté de l'extension .han, peut être édité avec un éditeur de texte quelconque (textedit,nedit,xemacs,vi,etc...). Si votre machine est dotée de nedit, vous pouvez accéder au fichier *.han (situé dans le répertoire anatomy), en cliquant sur l'oeuil correspondant.
Ce fichier a le format suivant :
sequence: inversion recovery
background: mean: 1
gray: mean: 25 sigma: 5
white: mean: 41 sigma: 4
Il vous suffit de spécifier une estimation grossière de la moyenne et de l'écart type des matières grises et blanches, puis de créer un fichier indiquant qu'il ne faut plus modifier cette analyse ultérieurement avec la commande:
touch nobias_dionysos.han.loc
Vous pouvez obtenir le même résultat avec la procédure Validation Nobias Histo analysis en mettant validation à Lock (+ execute)
Nous essaierons de simplifier cette interaction le plus rapidement possible...
Pour plus d'info:
Vip Histogram Analysis
Echec de la correction du biais
Pour évaluer si un échec de la correction de biais pourrait être à l'origine d'un échec de l'analyse d'histogramme, voire d'un échec du processus de segmentation (si une zone de l'image est trop claire ou trop sombre par rapport au reste, elle risque d'être éliminée), vous pouvez accéder à une visualisation en cliquant sur l'oeuil adéquat.
Pour apprendre à interpréter la visualisation: Validation Bias Correction from T1 mri.
Pour plus d'information sur la méthode de correction de biais :
Vip Bias Correction
Si tout vous semble correct, vous pouvez l'indiquer à brainvisa avec la procédure Validation Bias Correction from T1 MRI en mettant validation à Lock (+ execute)
mri: Raw T1 MRI ( entrée )
brain_mask: T1 Brain Mask ( sortie )Un masque binaire du cerveau
variant: Choice ( input )Une variante de la procédure Vip Get Brain
help: Choice ( input )Différentes visualisations auxiliaires
mri_corrected: IRM T1 Biais Corrigé ( entrée )L'image précédente corrigée du biais
histo_analysis: Analyse d'histogramme ( entrée )Statistiques sur les matières grises et blanches (fichier texte)
Commissure_coordinates: Commissure coordinates ( optional, entrée )
lesion_mask: Volume 3D ( optional, entrée )
first_slice: Entier ( input )Nombre de coupes à effacer au début
last_slice: Entier ( input )Nombre de coupes à effacer à la fin
Toolbox : Morphologist
Niveau d'utilisateur : 0
Identifiant :
AnaT1toBrainMaskCorrectionNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/correction/AnaT1toBrainMaskCorrection.pySupported file formats :
mri :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagebrain_mask :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imagemri_corrected :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagehisto_analysis :Analyse d'histogramme, Analyse d'histogrammeCommissure_coordinates :Commissure coordinates, Commissure coordinateslesion_mask :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 image