Validation de Vip Split Brain
Cet outil fusionne la segmentation avec l'IRM corrigée du biais de manière à simplifier la vérification du résultat. La visualisation obtenue est la suivante:
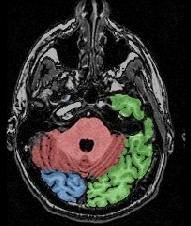
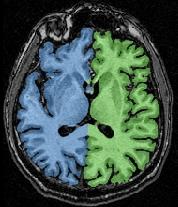
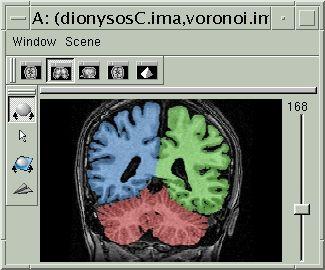
Si tout vous semble correct, vous pouvez l'indiquer à brainvisa en mettant validation à Yes (+ execute). Cette commande crée le fichier voronoi.loc. S'il existe, la segmentation ne sera plus jamais effectuée, même par la procédure Correction Split Brain from Brain Mask
split_mask: Séparation du masque du cerveau ( entrée )
mri_corrected: IRM T1 Biais Corrigé ( entrée )Le résultat de la correction de biais
validation: Choice ( input )validation de la segmentation
Toolbox : Morphologist
Niveau d'utilisateur : 0
Identifiant :
AnaSplitBrainfromBrainMaskValidationNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/validation/AnaSplitBrainfromBrainMaskValidation.pySupported file formats :
split_mask :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagemri_corrected :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 image