


Crée un maillage d'un ou deux hémisphères
Cette procédure crée un maillage de chacun des hémisphères cérébraux. Elle inclue un prétraitement qui ouvre les sillons afin de faciliter la lecture de la surface corticale.
Le maillage de cette nouvelle version est sphérique, ce qui autorise le gonflement pour le déplisser.
Side: Choice ( input )Gauche, Droit, ou les Deux Hémisphères
mri_corrected: IRM T1 Biais Corrigé ( entrée )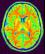
split_mask: Séparation du masque du cerveau ( entrée )
left_hemi_cortex: Left CSF+GREY Mask ( entrée )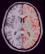
right_hemi_cortex: Right CSF+GREY Mask ( entrée )
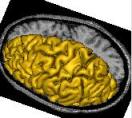
left_hemi_mesh: Left Hemisphere Mesh ( sortie )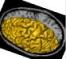
right_hemi_mesh: Right Hemisphere Mesh ( sortie )
fix_random_seed: Booléen ( input )
Toolbox : Morphologist
Niveau d'utilisateur : 2
Identifiant :
GetSphericalHemiSurfaceNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/segmentation/GetSphericalHemiSurface.pySupported file formats :
mri_corrected :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagesplit_mask :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imageleft_hemi_cortex :gz compressed NIFTI-1 image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIS image, JPEG image, MINC image, NIFTI-1 image, SPM image, TIFF image, TIFF(.tif) image, gz compressed MINC image, gz compressed NIFTI-1 imageright_hemi_cortex :gz compressed NIFTI-1 image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIS image, JPEG image, MINC image, NIFTI-1 image, SPM image, TIFF image, TIFF(.tif) image, gz compressed MINC image, gz compressed NIFTI-1 imageleft_hemi_mesh :GIFTI file, GIFTI file, Maillage MESH, MNI OBJ mesh, PLY mesh, Maillage TRIright_hemi_mesh :GIFTI file, GIFTI file, Maillage MESH, MNI OBJ mesh, PLY mesh, Maillage TRI