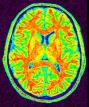

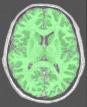



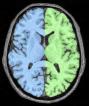
Sépare le masque du cerveau en trois parties : les hémisphères cérébraux et le reste (cervelet/tronc)
Cette procédure vise à séparer les hémisphères et à éliminer le cervelet et une partie du tronc cérébral de manière à accéder aux faces internes et basses du cortex.
Une érosion est appliquée à un masque de la matière blanche afin de la casser au niveau du corps calleux et du tronc cérébral. On récupère alors 3 graines correspondant aux hémisphères et au cervelet. Ces graines sont alors amenées à croître, dans un premier temps au sein de la matière blanche, puis au sein de la matière grise, de manière à retrouver les formes des hémisphères.
brain_mask: T1 Brain Mask ( entrée )
t1mri_nobias: IRM T1 Biais Corrigé ( entrée )
histo_analysis: Analyse d'histogramme ( entrée )
commissure_coordinates: Commissure coordinates ( optional, entrée )
use_ridges: Booléen ( input )
white_ridges: T1 MRI White Matter Ridges ( entrée )
use_template: Booléen ( input )
split_template: Hemispheres Template ( entrée )
mode: Choice ( input )
variant: Choice ( input )
bary_factor: Choice ( input )entre 0 et 1.
mult_factor: Choice ( optional, input )
initial_erosion: Réel ( input )
cc_min_size: Entier ( input )
split_brain: Séparation du masque du cerveau ( sortie )
fix_random_seed: Booléen ( input )
Toolbox : Morphologist
Niveau d'utilisateur : 0
Identifiant :
SplitBrainNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components/SplitBrain.pySupported file formats :
brain_mask :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imaget1mri_nobias :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagehisto_analysis :Analyse d'histogramme, Analyse d'histogrammecommissure_coordinates :Commissure coordinates, Commissure coordinateswhite_ridges :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagesplit_template :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagesplit_brain :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 image