Corrige le biais spatial d'une IRM
Cette procédure applique un traitement de l'ordre de la restauration de tableau...
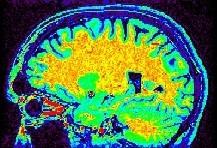
Alors qu'on se laisse facilement aller à imaginer qu'il existe une correspondence univoque entre l'intensité d'un voxel IRM, et une propriété ésotérique du cube de tissu sous-jacent, il faut se garder de succomber à cette tentation. Il suffit d'observer rapidement une image anatomique standard (pondérée en T1) avec une table de couleur arc-en-ciel pour s'en convaincre:
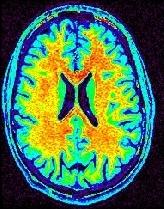
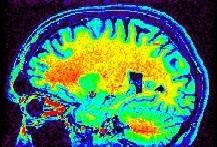
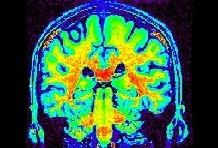
Certaines portions de la matière blanche sont rouges, d'autres jaunes voire vertes. Il semble donc que la correspondence évoquée ci-dessus dépende largement de l'endroit où on se situe dans le champ de vue. En général, le centre de l'antenne est plus clair, mais ce n'est pas systématique. Lorsqu'on se contente de regarder une coupe de cerveau en niveaux de gris, comme en radiologie conventionnelle, ce défaut ne saute pas aux yeux. La vision humaine est en effet particulièrement efficace lorsqu'il s'agit de corriger ce type de dérive spatiale. Cette capacité n'est d'ailleurs pas étrangère au fait que nous considérions généralement la couleur des objets comme uniforme malgré des variations d'illumination. Malheureusement, ce potentiel n'a pas poussé les physiciens à chercher à corriger ce défaut intrinsèque aux acquisitions. La vision artificielle ayant encore beaucoup de progrés à réaliser, ces variations spatiales d'intensité lui posent beaucoup plus de problèmes qu'au radiologue... Cette procédure cherche donc à restaurer l'image avant de passer la main aux procédures de segmentation proprement dites.
Le phénomène physique à l'origine de ces dérives spatiales dépend malheureusement de la forme de la tête du sujet. Il n'est donc pas possible d'appliquer une correction systématique après évaluation du problème sur un fantôme. Il faut donc estimer pour chaque nouvelle image la correction à effectuer. La démarche adoptée consiste à optimiser un critère mesurant la qualité de l'image restaurée. Ce critère est simplement une mesure de dispersion des intensités, l'entropie de leur distribution. Une entropie faible correspondra à une image où chaque classe de tissu est représentée par un pic étroit de l'histogramme. Lorsque l'image est biaisée spatialement, ces pics s'élargissent et l'entropie augmente.
Le modèle le plus simple que nous puissions considérer en ce qui concerne l'origine des intensités observées est le suivant:
L'intensité observée en un point x est supposée correspondre au produit d'une intensité intrinsèque au tissu I et d'un biais spatial F, auquel s'ajoute un bruit N propre à la RMN. Des considérations sur les multiples origines du biais nous conduisent à faire l'hypothèse qu'il est relativement régulier spatialement. Pour restaurer l'image O, nous cherchons donc à estimer le biais F, en supposant que le champ correctif Fc doit maximiser notre critère de qualité de l'image intrinsèque. Le champ correctif optimal est alors celui qui minise la fonction:
Le champ optimal réalise un compromis entre le critère de qualité (l'entropie S(FcO)), la régularité du champ (R(Fc), une somme d'écarts quadratique entre voisins), et une distance à l'image initiale (M(FcO) qui écarte la solution triviale correspondant à une image intrinsèque constante). Les constantes K pondèrent l'importance de ces trois termes. Cette fonction est minimisée par recuit simulé. La procédure de minimisation inclus en outre une approche multigrille qui permet de relacher la contrainte de régularité sur le champ. Il nous parait en effet probable que le champ correctif idéal contienne de fortes variations locales correspondant aux discontinuités du T1 des tissus. Pour l'image ci-dessus, la solution trouvée est la suivante:
L'image intrinsèque:
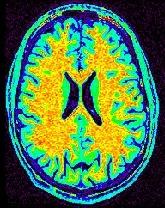
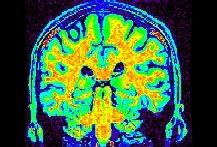
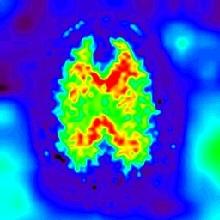
Le champ F:
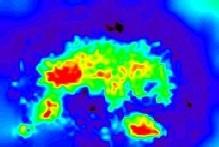
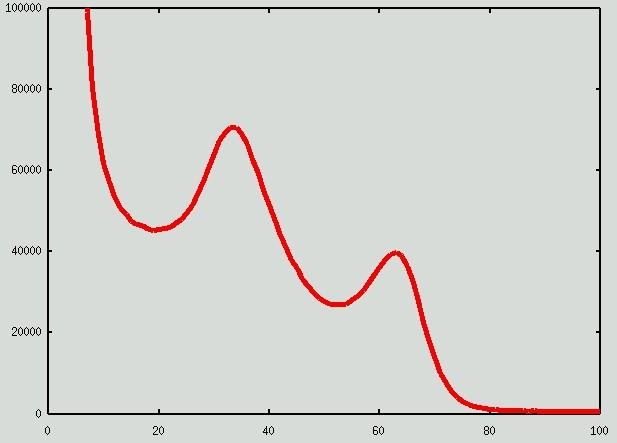
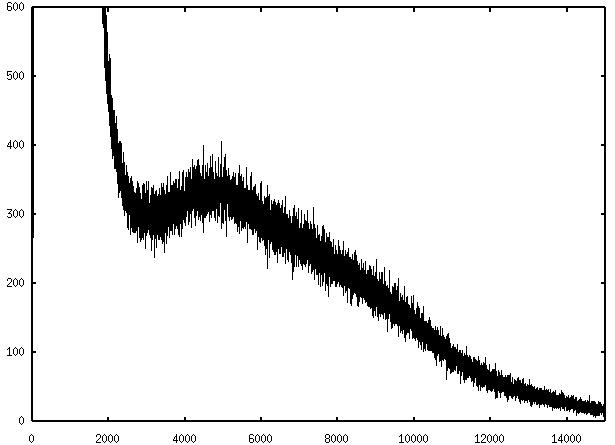
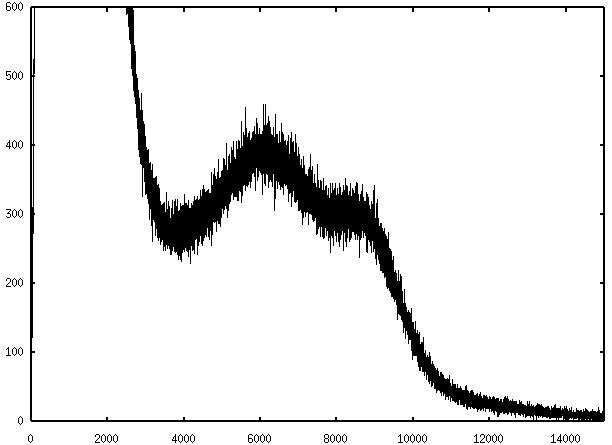
L'histogramme de l'image initiale O:
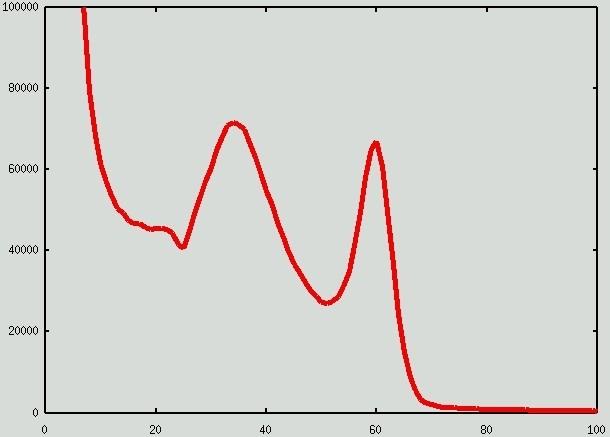
L'histogramme de l'image restaurée I:
Les principaux paramètres à modifier si vous rencontrez des problèmes avec vos images correspondent aux contraintes de régularité sur le champ:
- Si le contraste gris/blanc de vos images est faible, augmentez l'importance de cette contrainte (field_rigidity);
- Si votre antenne conduit à une dérive très importante dans la direction z (l'axe de l'aimant), diminuez la contrainte dans cette direction (zdir_multiply-regul entre 0.1 et 1);
- Si vos intensités varient d'une coupe à la suivante, passez en mode 2D (dim_rigidity).
Si vos images proviennent d'un imageur à haut champ, il est probable que le biais spatial soit très important. Dans ce cas il peut être nécessaire de diminuer la régularité du champ. Vous comprenez ainsi qu'une image avec un fort biais et un faible contraste peut s'avérer difficile à corriger... Cette procédure fonctionne néanmoins même lorsque le biais mélange complètement les pics gris et blanc de l'histogramme:
Exemple avec une image issue d'un imageur 3T:
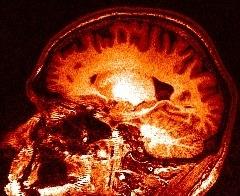
Le champ estimé:


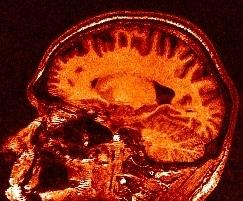
L'image restaurée:
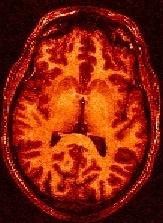
Pour plus d'information :
Entropy minimization for automatic correction of intensity non uniformity, J.-F. Mangin, MMBIA (Math. Methods in Biomed. Image Analysis), Hilton Head Island, South Carolina, IEEE Press 162-169, 2000
Vous pouvez également essayer la commande ligne:
VipBiasCorrection -help
mri: IRM T1 ( entrée )
une image quelconque (pas nécessairement T1)
mri_corrected: IRM T1 Biais Corrigé ( sortie )
l'image corrigée
field_rigidity: Réel ( input )pondération de la contrainte de rigidité du champ
write_field: Choice ( input )Ecriture du champ correctif
dim_rigidity: Choice ( input )2D rend les coupes indépendantes
sampling: Réel ( input )La taille en mm d'un bloc constant pendant l'optimisation
ngrid: Entier ( input )Nombre de niveaux de résolutions du champ
geometric: Réel ( input )raison de la décroissance de température du recuit simulé
nIncrement: Choice ( input )nombre de transition (x2) pour l'échantilloneur de Gibbs
init_temperature: Réel ( input )Température initiale
increment: Réel ( input )multiplicatif pour l'évolution du champ
init_amplitude: Réel ( input )multiplicatif, initialisation du champ
zdir_multiply_regul: Réel ( input )multiplicatif
Toolbox : Morphologist
Niveau d'utilisateur : 2
Identifiant :
VipBiasCorrectionNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/segmentation/VipBiasCorrection.pySupported file formats :
mri :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagemri_corrected :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 image