A pipeline to process Connectomist data of a subject into an individual connectivity matrix compatible with Constellation requirements.
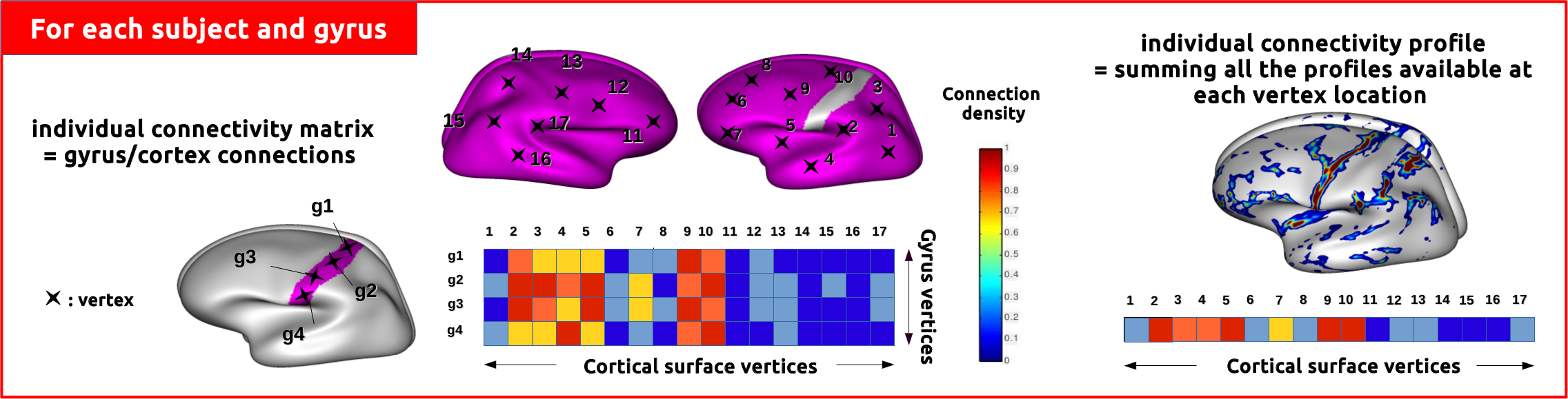
A complete pipeline: an intra_subject chain which builds the individual connectivity matrices for a given input "patch" (gyrus, or large region) of the brain. The preprocessings are done using FreeSurfer for the anatomical data, and Connectomist for the diffusion data.
The method implemented here is described in:
S. Lefranc, P. Roca, M. Perrot, C. Poupon, D. Le Bihan, J.-F. Mangin, and D. Rivière. Groupwise connectivity-based parcellation of the whole human cortical surface using watershed-driven dimension reduction. Medical Image Analysis, 30:11-29, 2016. [bibtex-entry]
To parcellate the whole brain, this process has to be iterated over all gyri.
Usage example
regions_nomenclature: /casa/build/share/brainvisa-share-4.6/nomenclature/translation/nomenclature_desikan_freesurfer.txtoutputs_database: /my/path/brainvisa_dbstudy_name: studyAmethod: averaged approachregion: lh.inferiorparietalsubject_indir: /my/path/connectomist_db/B1500/StreamlineProbabilistic/aQBI/27seeds/001individual_white_mesh: /my/path/freesurfer_db/001/surf/bh.r.aims.white.giidw_to_t1: /my/path/connectomist_db/B1500/StreamlineProbabilistic/aQBI/27seeds/001/dw_to_t1.trmregions_parcellation: /my/path/freesurfer_db/group_analysis/average_group/average_brain/bh.annot.averagebrain.giiregions_selection: Customkeep_regions: 'lh.unknown' 'lh.bankssts' 'lh.inferiorparietal'fiber_tracts_format: bundlesmin_fibers_length: 20.0max_fibers_length: 500.0smoothing: 3.0kmax: 12normalize: Trueerase_matrix: True
regions_nomenclature: Nomenclature ROIs File ( entrée )Nomenclature of the cortical parcellation used to partition the study.
Example : Freesurfer Desikan_Killiany Atlas
outputs_database: Choice ( input )Une base de données BrainVisa est obligatoire pour l'autocomplétion et l'écriture des fichiers de ce process. Sans celle-ci, aucune donnée pourra être générée. (voir la documentation pour ajouter une base de données dans la configuration de BrainVisa). Ce paramètre récupère l'ensemble des bases de données ayant une ontologie brainvisa présente dans votre configuration, vous n'avez plus qu'à choisir celle qui vous convient pour écrire les fichiers générés.
study_name: OpenChoice ( input )Ce paramètre obligatoire est libre et permet de donner un nom au pipeline. La valeur de "study_name" sera intégrée à chaque nom de fichier de sortie ainsi qu'à l'arborescence de la base de données qui récupère les fichiers de sortie.
method: Choice ( input )Deux méthodes sont proposées:
(1) "averaged approach" pour obtenir un unique résultat de groupe.
(2) "concatenated approach" pour obtenir des résultats individuels à l'échelle du groupe.
Incidences du choix de la méthode sur le paramètre "ROIs_nomenclature":
(1) "averaged approach" , dans le cadre de cette approche le paramètre "ROIs_nomenclature" doit être un fichier représentant la segmentation moyenne du groupe de sujets
(2) "concatenated approach", le paramètre "ROIs_nomenclature" est une segmentation individuelle.
region: OpenChoice ( input )The study region based on regions_nomenclature file.
subject_indir: Subject ( entrée )Ceci doit être le répertoire du sujet dans une base organisée à la facon Connectomist, où les fichiers de tractographie de fibres peuvent être trouvés.
individual_white_mesh: White Mesh ( entrée )Freesurfer white-grey interface of the cortex.
Should not be inflated.
dw_to_t1: Transform T2 Diffusion MR to Raw T1 MRI ( entrée )Affine spatial transformation to get the T1 MRI space from the dMRI diffusion (and tracts) space.
regions_parcellation: ROI Texture ( entrée )Cortical parcellation used to partition the study.
Example : Freesurfer Desikan_Killiany Atlas (?h.aparc.annot).
regions_selection: Choice ( input )This parameter can be used to exclude some regions from the data analysis. This is useful to study the specific connectivity between a given set of regions, or to exclude the initial patch region.
keep_regions: ListOf( OpenChoice ) ( input )Kept regions for the data analysis.
fiber_tracts_format: Choice ( input )Différents formats de faisceaux de fibres sont pris en charge. Les formats acceptés sont les suivants.
(1) Format de fichier TrackVis avec .trk comme extension de nom de fichier.
(2) Format de fichiers Connectomist avec .bundles comme extension de nom de fichier.
min_fibers_length: Réel ( input )A filtering parameter to exclude low length fibers. Default to 20mm.
max_fibers_length: Réel ( input )A filtering parameter to exclude long length fibers. Default to 500mm.
smoothing: Réel ( input )Degré du smoothing en millimètre.
kmax: Entier ( input )Maximal number of clusters used to parcellate the study region.
Default to 12.
normalize: Booléen ( input )By default the connectivity matrices values are normalized to balance any spurious weighting effects due to more connected regions or subjects, or to the tractography algorithm (number of seeds...). But sometimes we also like to see the raw connectivity matrix: in that case, uncheck this normalization.
erase_smoothed_matrix: Booléen ( input )
Toolbox : Constellation
Niveau d'utilisateur : 1
Identifiant :
constel_individual_pipelineNom de fichier :
brainvisa/toolboxes/constellation/processes/individual_pipelines/constel_individual_pipeline.pySupported file formats :
regions_nomenclature :Text file, Text filesubject_indir :Répertoire, Répertoireindividual_white_mesh :GIFTI file, GIFTI file, Maillage MESH, MNI OBJ mesh, PLY mesh, Maillage TRIdw_to_t1 :Transformation matrix, Transformation matrixregions_parcellation :GIFTI file, GIFTI file, Texture