Validation of Vip Bias Correction
Pour évaluer si un échec de la correction de biais pourrait être à l'origine d'un échec de l'analyse d'histogramme, voire d'un échec d'un des processus de segmentation ultérieurs (lorsque une zone de l'image est trop claire ou trop sombre par rapport au reste, elle risque d'être éliminée), vous pouvez comparer l'image corrigée avec l'image initiale, voire avec le résultat de la segmentation, en utilisant simultanément d'autres briques de visualisation:
Visualiser la correction de biais
L'image IRM brute et sa version corrigée du biais vont apparaÎtre dans Anatomist dotées d'une table de couleur arc-en-ciel. En jouant sur le réglage de la table de couleur, vous pouvez prendre conscience du biais dans l'image initiale (en général, l'image est plus claire au centre du champ de vue) :
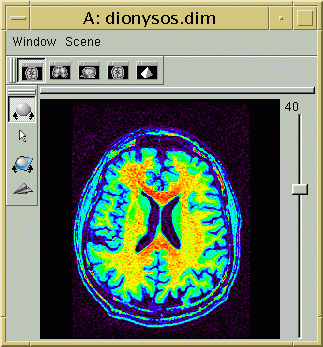
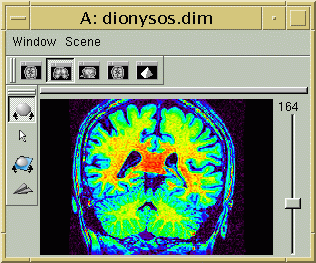
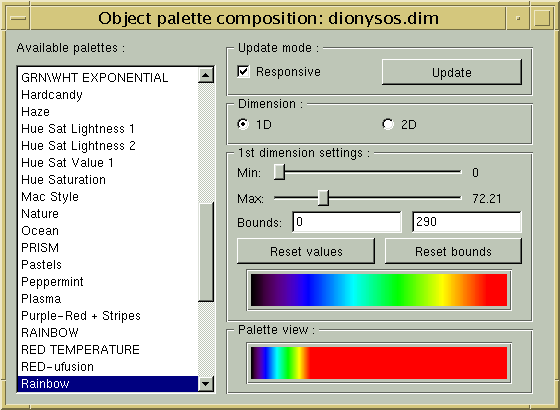
En revanche, cet effet doit avoir disparu après la correction. Pour s'en convaincre, il faut pouvoir régler la table de couleur de manière à faire apparaître la matière blanche comme un mélange régulier de jaune et de rouge :
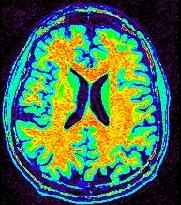
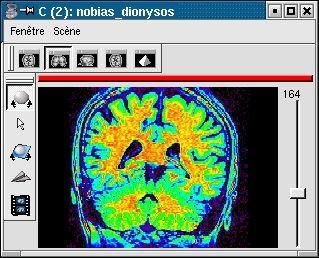
Si tout vous semble correct, vous pouvez l'indiquer à brainvisa en mettant validation à Lock (+ execute). Cette commande crée le fichier nobias_dionysos.loc. S'il existe, la correction de biais, qui est assez longue, ne sera plus jamais effectuée.
mri_corrected: T1 MRI Bias Corrected ( input )
T1mri: Raw T1 MRI ( input )
validation: Choice ( input )
Toolbox : Morphologist
User level : 0
Identifier :
AnaT1toBiasCorrectionValidationSupported file formats :
mri_corrected :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Directory, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imageT1mri :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Directory, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 image