




Crée le graphe des plis corticaux.
Cette procédure transforme l'image IRM en une structure qui résume l'essentiel de l'agencement des plissements du cortex. Il s'agit d'extraire de l'énorme quantité d'informations contenue dans les intensités de l'image volumique une représentation simplifiée mais suffisante pour procéder à la reconnaissance des sillons ou des racines sulcales. Cette nouvelle représentation est un graphe dont les noeuds correspondent à des plissements corticaux élémentaires, et dont les relations indiquent l'agencement relatif de ces plissements. Cette nouvelle représentation permet de manipuler les formes du cortex d'une manière abstraite plus adaptée à certaines techniques de reconnaissance des formes qui tentent de s'inspirer de la démarche du neuro-anatomiste: les entités manipulées ne seront plus des voxels et leurs intensités, mais des plissements corticaux, leurs tailles et leurs orientations, leurs connexions éventuelles...
Voici par exemple la représentation sous forme de graphe obtenue pour un sillon relativement simple:
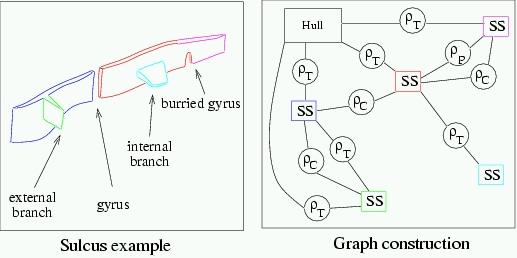
Le sillon est segmenté en 5 plis élémentaires qui correspondent aux noeuds du graphe (les carrés). L'enveloppe externe du cerveau apparaît également comme un noeud du graphe. Enfin, les relations qui relient ces noeuds (les ronds) représentent différentes informations topographiques:
- T correspond à une jonction entre deux plis ou entre un pli et l'enveloppe du cerveau ;
- C correspond à une notion de voisinage géodésique à l'enveloppe du cerveau : on peut passer d'un pli à son voisin en passant par l'extérieur du cortex sans traverser un autre pli. Ce voisinage correspond aux adjacences d'un diagramme de Voronoï géodésique à l'enveloppe du cerveau calculé pour les jonctions des plis avec cette enveloppe. Ce diagramme est une parcellisation de l'enveloppe en fonction de la jonction la plus proche. Chaque pli élémentaire induit ainsi une zone d'influence autour de lui. Deux plis sont reliés si ces zones se touchent ;
- P correspond à la notion anatomique de "pli de passage" décrite au siècle dernier. Il s'agit d'un gyrus enfoui au fond d'un sillon qui peut émerger chez certains individus et par là même interrompre le sillon. La présence possible d'un gyrus enfoui est détectée à partir de critères de profondeur et de courbure de la surface corticale.
La construction du graphe implique toute une chaîne de traitement:
Pour le lecteur névrosé qui veut vraiment en savoir encore plus:
- La détection d'une interface gris/blanc dotée de la topologie sphérique du cortex. La technique utilisée part du masque d'un hémisphère fourni par Ana Split Brain From Brain Mask:
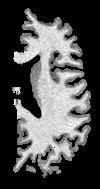
Ce masque est placé dans une boîte parallélépipédique dont l'interface interne se déforme pour atteindre l'interface gris blanc et dont l'interface externe se déforme pour atteindre l'enveloppe de l'hémisphère:

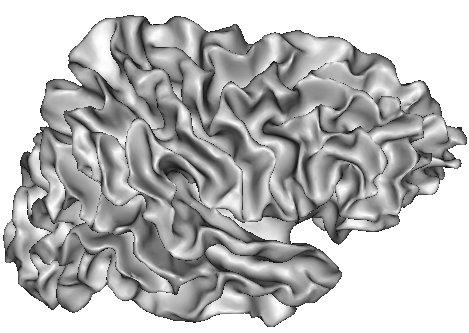
Le maillage de l'interface gris-blanc permet alors d'obtenir une représentation du cortex de topologie sphérique qui sera gonflée par d'autres briques de Brainvisa à des fins de visualisation:

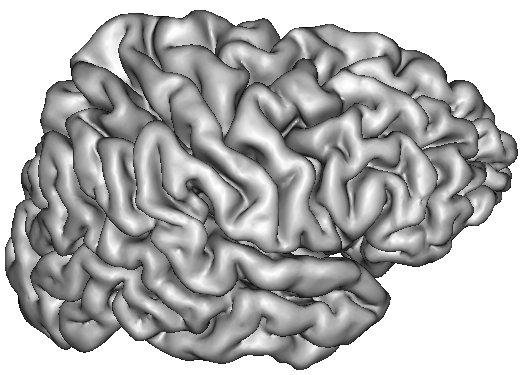
Une dilatation de cette interface vers l'extérieur permet également de réaliser des rendus de la surface du cortex relativement lisibles car les sillons sont ouverts (Ana Get Openen Hemi Surface) :

- L'étape suivante consiste à squelettiser l'objet blanc évoqué ci-dessus. Il s'agit en fait d'une érosion qui rogne cet objet de l'intérieur jusqu'à ce qu'il devienne sans épaisseur. Le squelette final ressemble à un moule en négatif du cerveau, ou aux feuillets qui émanent parfois de la cosse des noix de Grenoble et qui remplissent ses plissements.
Imaginez qu'un hémisphère cérébral soit fendu en deux, et que la matière blanche soit retirée du résultat. Réalisé virtuellement, cette technique permet de visualiser les plissements corticaux de dessous:
Il est alors facile de comprendre l'effet de la squelettisation:
Le squelette est donc constitué de l'enveloppe du cerveau et de nombreuses surfaces médiales aux plissements:
L'érosion pratiquée pour réaliser la squelettisation n'est en fait pas uniforme. En effet, pour s'affranchir d'éventuelles différences d'épaisseur de cortex de part et d'autre du sillon, et pour maîtriser la profondeur finale du squelette, l'image en niveau de gris est utilisée pour définir la localisation préférentielle pour le squelette. Si on imagine que le niveau de gris correspond à une altitude, la matière blanche correspond aux crêtes des montagnes, et le liquide au fond des crevasses. Pour imposer la localisation du squelette au fond des crevasses, nous utilisons un "détecteur de crevasses"... Pour les initiés, il s'agit de la courbure moyenne des isophotes de l'image volumique:
Ce détecteur n'est pas parfait, car le fond des crevasses peut contenir des trous. Le processus d'érosion utilisé permet de combler ces trous (pour les initiés, il s'agit s'une érosion homotopique). Il procède comme la montée des eaux qui détruit peu à peu un chateau de sable. L'altitude du chateau en chaque point correspond à la réponse du détecteur de crevasse. Chaque vague va enlever un peu de sable au niveau des murs extérieurs. Petit à petit, les murs vont s'effondrer et passer sous le niveau de l'eau. Les parties les plus élevées du chateau seront les dernières à s'effondrer. Lorsque deux fronts de vagues se rencontrent, une portion de squelette est édifiée:
Le détecteur de crevasse:
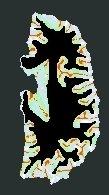
Le squelette final (les couleurs correspondent à une classification topologique des jonctions et des fonds de sillons):

Cette visualisation en coupe est d'ailleurs plutôt perturbante, car elle ne permet pas d'appréhender le fait que ce squelette est toujours doté d'une topologie sphérique (l'homotopie d'une sphère en fait...)- Etape suivante (pour ceux qui n'ont pas encore craqué...).
Le but de cette squelettisation était d'atteindre un stade où il soit facile d'extraire un à un les plissements corticaux élémentaires. Il suffit de casser le squelette au niveau des jonctions pour obtenir le puzzle que nous désirons. Cette opération est réalisée à partir d'une technique de topologie discrète qui s'interprète de la facon suivante. Imaginez qu'on colle deux aimants de part et d'autre d'une portion du squelette :
Tous les points atteignables en faisant glisser les aimants sans les décoller seront attribués au même pli élémentaire. Dans le jargon de la topologie discrète, un tel ensemble s'appelle une surface simple. Cette opération est appliquée à l'ensemble des points du squelette qui séparent le fond en deux composantes connexes (ce qui élimine le fond des sillons et certaines jonctions (il n'existe malheureusement pas de détecteur exhaustif de points de jonctions à l'heure actuelle, ce qui aurait le mérite de simplifier les choses...)). Il s'agit des points jaunes :
Zoom sur un morceau de squelette:
On ne garde plus que l'enveloppe du cerveau en orange :
La segmentation:
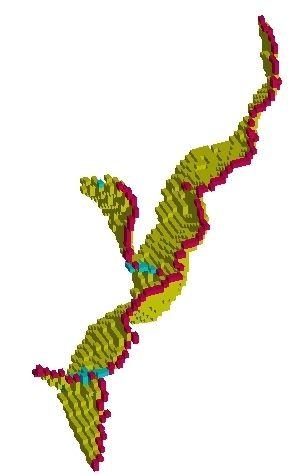
- Dernière étape, la détection des gyri enfouis putatifs (et oui je l'ai placé...). Il s'agit de doter notre représentation du cortex des informations nécessaires pour étudier la notion de racine sulcale sur laquelle je ne vais pas m'étendre ici. Nous tentons de définir un modèle des plissements corticaux fondé sur des unités élémentaires insécables susceptibles de correspondre aux premiers plissements corticaux apparaissant in utero. Ces efforts visent à pallier les faiblesses des descriptions actuelles en ce qui concerne la gestion des interruptions des sillons, phénomène qui a même été décrit pour le sillon central:
Nous cherchons donc à casser le sillon central au niveau du "pli de passage" qui le sépare en deux racines sulcales, ce qui donne, vu du dessous :
Les surfaces simples sont donc susceptibles d'être segmentées en plusieurs morceaux lorsque des variations de profondeur ou de courbure gausienne semblent indiquer la présence d'un gyrus enfoui. Je ne vais pas décrire en détail la procédure utilisée, car elle est encore loin d'être satisfaisante. Nos efforts actuels pour l'améliorer sont décrits dans:
A mean curvature based primal sketch to study the cortical folding process from antenatal to adult brain.
A. Cachia, J.-F. Mangin, D. Rivière, N. Boddaert, A. Andrade, F. Kherif, P. Sonigo, D. Papadopoulos-Orfanos, M. Zilbovicius, J.-B. Poline, I. Bloch, F. Brunelle, and J. Régis.
In MICCAI, Utrecht, The Netherlands, LNCS-2208, pages 897--904. Springer Verlag, 2001.
Voici quelques images pour se faire une idée du processus actuel sur la fissure calloso marginale :
LA profondeur géodésique :
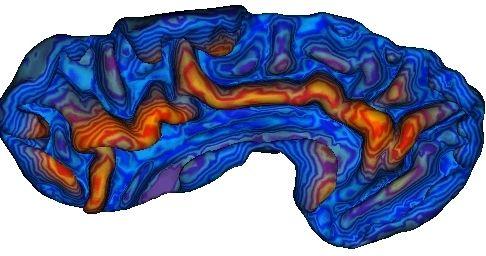
La courbure gausienne de la surface corticale, qui indique les zones de la forme d'une selle de cheval susceptibles de correspondre à un gyrus enfoui :
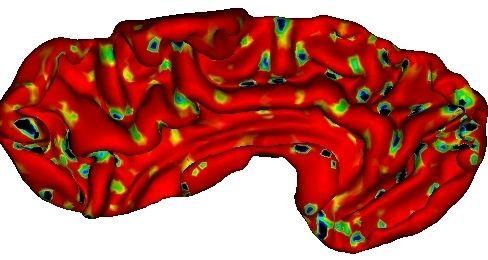
La parcellisation correspondant aux gyri enfouis :
Le squelette :
La segmentation finale qui combine la notion de surface simple et de gyrus enfoui :
Oui, je sais, ca fait beaucoup de morceaux...
From 3D magnetic resonance images to structural representations of the cortex topography using topology preserving deformations.
J.-F. Mangin, V. Frouin, I. Bloch, J. Regis, and J.Lopez-Krahe.
Journal of Mathematical Imaging and Vision, 5(4):297--318,1995.
Automatic recognition of cortical sulci using a congregation of neural networks.
D. Rivière, J.-F. Mangin, D. Papadopoulos-Orfanos, J.-M. Martinez, V. Frouin, and J. Régis.
In MICCAI, Pittsburgh, LNCS-1935, 40-49. Springer Verlag, 2000 (à paraître dans Medical Image Analysis, 2002).
Side: Choice ( input )travaille sur un ou deux hémisphères
mri_corrected: IRM T1 Biais Corrigé ( entrée )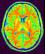
histo_analysis: Analyse d'histogramme ( entrée )
split_mask: Séparation du masque du cerveau ( entrée )
left_hemi_cortex: Left CSF+GREY Mask ( sortie )
right_hemi_cortex: Right CSF+GREY Mask ( sortie )détection de l'interface gris blanc
Lskeleton: Left Cortex Skeleton ( sortie )Squelette de l'objet précédent
Rskeleton: Right Cortex Skeleton ( sortie )
Lroots: Left Cortex Catchment Bassins ( sortie )Bassins versants d'une ligne de partage des eaux
Rroots: Right Cortex Catchment Bassins ( sortie )
Lgraph: Graphe de sillons corticaux ( sortie )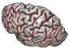
Rgraph: Graphe de sillons corticaux ( sortie )
Commissure_coordinates: Commissure coordinates ( entrée )
compute_fold_meshes: Choice ( input )
Toolbox : Morphologist
Niveau d'utilisateur : 2
Identifiant :
AnaComputeCorticalFoldArgNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/segmentation/AnaComputeCorticalFoldArg.pySupported file formats :
mri_corrected :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagehisto_analysis :Analyse d'histogramme, Analyse d'histogrammesplit_mask :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imageleft_hemi_cortex :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageright_hemi_cortex :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageLskeleton :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageRskeleton :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageLroots :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageRroots :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageLgraph :Graph and data, Graph and dataRgraph :Graph and data, Graph and dataCommissure_coordinates :Commissure coordinates, Commissure coordinates