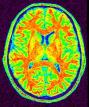

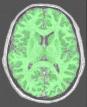



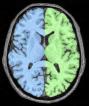
Sépare le masque du cerveau en trois parties : les hémisphères cérébraux et le reste (cervelet/tronc)
Cette procédure vise à séparer les hémisphères et à éliminer le cervelet et une partie du tronc cérébral de manière à accéder aux faces internes et basses du cortex.
Une érosion est appliquée à un masque de la matière blanche afin de la casser au niveau du corps calleux et du tronc cérébral. On récupère alors 3 graines correspondant aux hémisphères et au cervelet. Ces graines sont alors amenées à croître, dans un premier temps au sein de la matière blanche, puis au sein de la matière grise, de manière à retrouver les formes des hémisphères.
brain_mask: T1 Brain Mask ( entrée )
t1mri_nobias: IRM T1 Biais Corrigé ( entrée )
histo_analysis: Analyse d'histogramme ( entrée )
commissure_coordinates: Commissure coordinates ( optional, entrée )
use_ridges: Booléen ( input )
white_ridges: T1 MRI White Matter Ridges ( entrée )
use_template: Booléen ( input )
split_template: Hemispheres Template ( entrée )
mode: Choice ( input )
variant: Choice ( input )
bary_factor: Choice ( input )entre 0 et 1.
mult_factor: Choice ( optional, input )
initial_erosion: Réel ( input )
cc_min_size: Entier ( input )
split_brain: Séparation du masque du cerveau ( sortie )
fix_random_seed: Booléen ( input )
Toolbox : Morphologist
Niveau d'utilisateur : 0
Identifiant :
SplitBrainNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components/SplitBrain.pySupported file formats :
brain_mask :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imaget1mri_nobias :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagehisto_analysis :Analyse d'histogramme, Analyse d'histogrammecommissure_coordinates :Commissure coordinates, Commissure coordinateswhite_ridges :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagesplit_template :gz compressed NIFTI-1 image, Aperio svs, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, Ventana bif, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagesplit_brain :gz compressed NIFTI-1 image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIS image, JPEG image, MINC image, NIFTI-1 image, SPM image, TIFF image, TIFF(.tif) image, gz compressed MINC image, gz compressed NIFTI-1 image