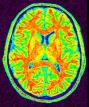

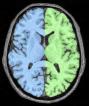



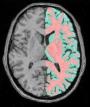
Classification Gris/Blanc
Cette brique propose une classification gris blanc de chacun des hémisphères cérébraux pour les personnes souhaitant tester l'influence des algorithmes de classification (SPM, outils du MNI, SHFJ...) sur le résultat final de la VBM...
L'approche que nous proposons traite séparément chacun des hémisphères, ce qui devrait permettre d'éviter des mélanges entre les faces médiales, ou entre les faces basses et le cervelet, au cours du lissage spatial préalable aux statistiques.
Une autre particularité de l'approche proposée : nous avons décidé d'écarter le cervelet de la classification dans la mesure où les problèmes de volume partiel sont à notre avis trop importants pour aboutir à un résultat correct (une classification floue pourrait résoudre ce problème à l'avenir).
mri_corrected: IRM T1 Biais Corrigé ( entrée )
histo_analysis: Analyse d'histogramme ( entrée )
split_mask: Séparation du masque du cerveau ( entrée )
edges: T1 MRI Edges ( entrée )
commissure_coordinates: Commissure coordinates ( entrée )
Side: Choice ( input )Gauche, Droit, ou les Deux Hémisphères
left_grey_white: Left Grey White Mask ( sortie )classification (gris=100, blanc=200)
right_grey_white: Right Grey White Mask ( sortie )classification (gris=100, blanc=200)
fix_random_seed: Booléen ( input )
Toolbox : Morphologist
Niveau d'utilisateur : 2
Identifiant :
GreyWhiteClassificationNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components_obsolete/segmentation/GreyWhiteClassification.pySupported file formats :
mri_corrected :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagehisto_analysis :Analyse d'histogramme, Analyse d'histogrammesplit_mask :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imageedges :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagecommissure_coordinates :Commissure coordinates, Commissure coordinatesleft_grey_white :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageright_grey_white :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 image