Spécification des commissures
Pour profiter de l'organisation fournie par Brainvisa lors de l'utilisation des routines de segmentation du SHFJ, il est préférable de s'appuyer sur cette brique qui permet de spécifier l'intersection des commissures avec le plan interhémisphérique, ainsi qu'un point supplémentaire de ce plan, de manière à permettre un passage dans le référentiel de Talairach. Ce référentiel permet aux routines d'analyse d'images diverses vérifications, ainsi qu'une initialisation fiable de certaines recherches. Si votre image a été normalisée avec SPM ou MRItotal (outil du MNI), spécifiez-le et vous échapperez à la corvée de sélection manuelle (mais vérifiez sur un exemple que tout se passe bien...)
Pour utiliser cette brique sur un sujet, il faut avoir préalablement importé l'IRM T1 du sujet dans la "base de données" de BrainVISA: ceci se fait à l'aide du traitement d'importation d'IRM anatomique.
Pour vérifier que Brainvisa a bien intégré l'existence de ce nouveau sujet, ouvrez la brique "Prepare Subject for Anatomical Pipeline".
Cliquez sur la loupe pour accéder a la liste des sujets pris en compte par Brainvisa.
Votre nouveau sujet doit apparaître dans la liste. Si votre base de donnéees contient beaucoup de sujets, vous pouvez procéder à divers tris à l'aide des boutons "protocole, voxel_size..." pour accéder plus facilement à celui qui vous intéresse.
Tout se passe bien; sélectionnez votre sujet:
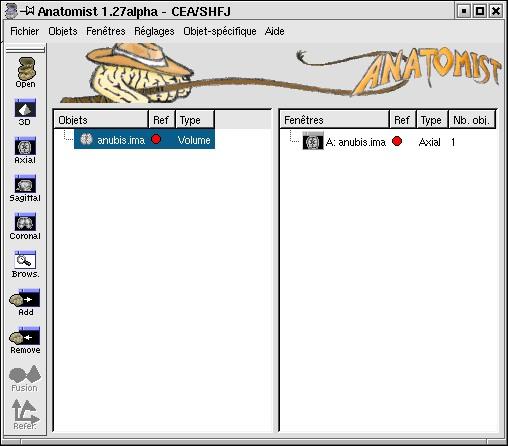
l'oeil de Brainvisa est maintenant activable, ce qui signifie qu'il est capable d'envoyer l'IRM à Anatomist.
Cliquez sur l'oeil, pour le vérifier...
Vous devez maintenant voir les fenêtres suivantes:
Il vous reste à spécifier la localisation des commissures. Si votre image a été normalisée à partir d'une procédure standard répertoriée par Brainvisa dans le choix "Normalised", sélectionnez la et exécutez la brique...
Les procédures gérées pour l'instant sont:
Il vous faut comprendre que si vous n'avez pas rééchantilloné votre image avec une bonne résolution spatiale (autour d'1mm), les processus de segmentation donneront de mauvais résultats, même si vous spécifiez les commissures manuellement... En effet, la résolution des plis corticaux est souvent de l'ordre du millimètre. En outre, si votre image est dotée de coupes épaisses, vous risquez de rencontrer d'autres problèmes...
- SHFJ from SPM: champ de vue minimal, resolution 1x1x1mm
- MNI from Mritotal: resolution 1x1x1mm
- Marseille from SPM: resolution 1x1x1mm et plus grand champ de vue
NB: il n'est pas nécessaire d'avoir un accord parfait en ce qui concerne la localisation des commissures. Les procédures de normalisation automatique ne prennent en effet pas spécifiquement en compte leur localisation... En cas de doute sur l'adéquation d'une de ces procédures à vos données, vous pouvez vérifier leur position à l'aide du visualiseur AC/PC (oeil).
Si tout vous semble convenable, vous pouvez itérer le traitement sur l'ensemble des sujets de votre protocole.
Si votre image n'est pas normalisée, Anatomist va vous aider à spécifier trois ou quatre points en "cliquant" sur l'image visualisée.
- Si le volume n'est pas déjà visible, cliquez une première fois sur l'icône d'édition associée au paramètre correspondant au point à entrer (AC, PC ou IP) (icône "anatomist"): l'image se charge et s'affiche dans Anatomist
- Positionnez une vue d'anatomist de manière à voir le point en question (en bougeant le curseur de coupe), et cliquez, sur la vue anatomist, pour amener le curseur sur le point voulu.
- quand la position est bonne, cliquez l'icône du point 3D (dans BrainVisa) pour reporter les coordonnées d'anatomist vers BrainVISA.
- Si vous n'êtes pas satisfait, vous pouvez réitérer l'opération en cliquant sur d'autres points: une nouvelle position remplace l'ancienne dans BrainVisa dès que l'on re-clique sur le bouton du paramètre BrainVISA.
Vous pouvez choisir les trois points en vous aidant d'une coupe sagittale si vous préférez:
- L'intersection de la commissure antérieure avec le plan inter-hémisphérique;
- L'intersection de la commissure postérieure avec le plan inter-hémisphérique;
- Un point quelconque du plan inter-hémisphérique qui ne soit pas alligné avec les précédents.
- Optionnellement, un point quelconque de l'hémisphère gauche. Il n'est pas obligatoire de spécifier ce point, il sert uniquement à détecter un éventuel problème d'orientation de l'image. Attention, les coupes axiales et coronales d'Anatomist sont par défaut en convention radiologique (droite et gauche inversées).
NB: Si le réglage automatique de la palette de couleur ne vous convient pas, vous pouvez le modifier en sélectionnant votre image dans la fenêtre de controle d'Anatomist, et en allant chercher "palette" dans le menu "objet-spécifique".
Commissure antérieure:
Commissure postérieure:
Point du plan interhémisphérique:
Point de l'hémisphère gauche (à droite sur une coupe axiale affichée en convention radiologique):
Vous devez avoir maintenant spécifié les 12 coordonnées:
Un dernier paramètre, allow_flip_initial_MRI, précise si vous autorisez ou non BrainVisa à réécrire l'image d'entrée s'il s'avère qu'elle a besoin d'être réorientée avant de passer par la moulinette de segmentation. En effet nos outils supposent que les images ont une orientation axiale et sont dans un repère radiologique et gauche. Si ce n'est pas le cas, l'étape de séparation des hémisphères a de fortes chances de rater, l'identification des sillons ne pourra pas aboutir à un résultat cohérent, et le lien avec un repère commun (Talairach) sera faux.
Il vous reste à exécuter la brique... Elle doit créer un fichier texte anubis.APC qui contient les coordonnées mentionnées.
NB: Il est possible d'utiliser les outils de segmentation du SHFJ sans créer ce fichier, à vos risques et périls. En revanche, les pipelines généraux vous seront interdits par mesure de sécurité...
T1mri: Raw T1 MRI ( entrée )
commissure_coordinates: Commissure coordinates ( sortie )
Normalised: Choice ( input )Une procedure de normalisation spatiale
Anterior_Commissure: Point3D ( optional, input )Position de la commissure antérieure (mm)
Posterior_Commissure: Point3D ( optional, input )PC (mm)
Interhemispheric_Point: Point3D ( optional, input )Un point du plan inter-hémisphérique (mm). Prenez-le assez loin de AC et PC de préférence (vers le haut du cerveau)
Left_Hemisphere_Point: Point3D ( optional, input )N'importe quel point de l'hémisphère gauche (pas trop près du plan inter-hémisphérique)
allow_flip_initial_MRI: Booléen ( input )Autorise ou empêche la réécriture de l'image d'entrée s'il est nécessaire de changer son orientation
reoriented_t1mri: Raw T1 MRI ( sortie )
remove_older_MNI_normalization: Booléen ( input )Ce paramètre contrôle l'effacement d'informations plus anciennes de normalisation dans l'espace du MNI, pour éviter des ambigüités entre des transformations contradictoires.
older_MNI_normalization: Transform Raw T1 MRI to Talairach-MNI template-SPM ( optional, entrée )Anciennes informations de normalisation vers l'espace du MNI. Elles seront juste effacées, si elles sont présentes, et si remove_older_MNI_normalization est activé.
Toolbox : Morphologist
Niveau d'utilisateur : 0
Identifiant :
preparesubjectNom de fichier :
brainvisa/toolboxes/morphologist/processes/segmentationpipeline/components/preparesubject.pySupported file formats :
T1mri :gz compressed NIFTI-1 image, Aperio svs, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, FreesurferMGH, FreesurferMGZ, GIF image, GIS image, Hamamatsu ndpi, Hamamatsu vms, Hamamatsu vmu, JPEG image, Leica scn, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, Sakura svslide, TIFF image, TIFF image, TIFF(.tif) image, TIFF(.tif) image, VIDA image, Ventana bif, XBM image, XPM image, Zeiss czi, gz compressed MINC image, gz compressed NIFTI-1 imagecommissure_coordinates :Commissure coordinates, Commissure coordinatesreoriented_t1mri :gz compressed NIFTI-1 image, BMP image, DICOM image, Répertoire, ECAT i image, ECAT v image, FDF image, GIF image, GIS image, JPEG image, MINC image, NIFTI-1 image, PBM image, PGM image, PNG image, PPM image, SPM image, TIFF image, TIFF(.tif) image, VIDA image, XBM image, XPM image, gz compressed MINC image, gz compressed NIFTI-1 imageolder_MNI_normalization :Transformation matrix, Transformation matrix